范德比尔特大学的一组研究人员发布了一项新的基准研究,旨在帮助科学家选择最有效的方法来分析空间转录组学(ST)数据。
该研究由生物医学工程和计算机科学助理教授周昕迈兹领导,评估了空间转录组学(ST)中的计算工具,这是一种用于绘制组织中基因表达模式同时保留空间背景的技术。这项研究最近发表在《基因组生物学》杂志上。
ST包括切片组织样本,并将其放置在带有空间索引条形码的特殊设计的载玻片上。当组织被处理时,组织中每个特定位置的核糖核酸(RNA)被这些条形码捕获。在对RNA进行测序后,数据可以映射回原始组织位置,使研究人员能够可视化某些基因在组织结构中表达的位置。
自2020年开始广泛使用以来,这项突破性的测序技术彻底改变了对复杂组织的理解。ST的应用包括癌症研究和神经科学,例如绘制大脑部分区域的基因表达以了解区域功能或疾病机制。
然而,用于分析ST数据的各种可用工具可能是压倒性的,这使得很难为特定的研究需求选择正确的方法。
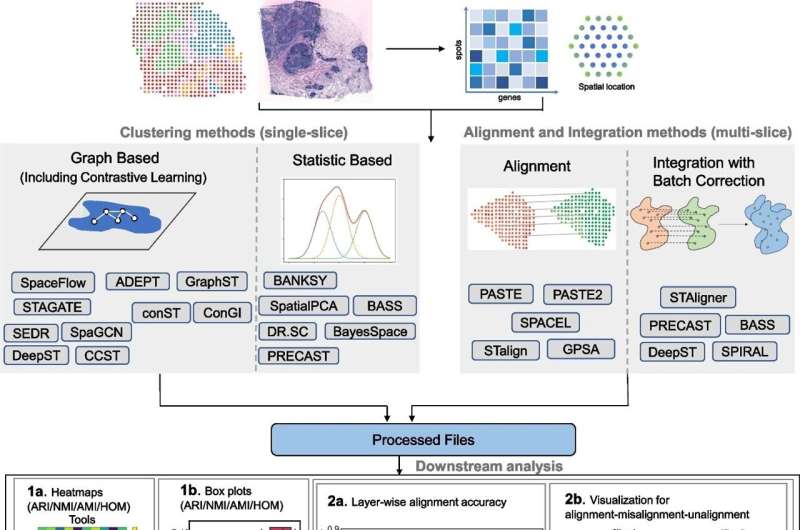
为了解决这个问题,范德比尔特大学的研究小组系统地比较了16种聚类方法、5种对齐方法和5种跨各种数据集的集成方法。他们的发现为研究空间转录组学的研究人员提供了实用的建议,帮助他们确定最符合他们研究要求的工具。
“我们的目标是为研究人员在空间转录组学分析中导航提供一个清晰易懂的指南,”周说,他是范德比尔特大学跨机构数据科学研究所的教师之一。“我们希望这项研究将成为这个快速发展领域的工作人员的有用资源。”
本文来自作者[佼得深]投稿,不代表思达普立场,如若转载,请注明出处:https://m.cdstartup.cn/zlan/202505-574.html


评论列表(4条)
我是思达普的签约作者“佼得深”!
希望本篇文章《基准研究助力科学家解析空间转录组学数据》能对你有所帮助!
本站[思达普]内容主要涵盖:国足,欧洲杯,世界杯,篮球,欧冠,亚冠,英超,足球,综合体育
本文概览: ...